Collaborations
- Complexe intégron/integrase : Dr Gopaul, Institut Pasteur (Paris)
- Modélisation : Dr Tarrat, CEMES (Toulouse)
- Aptamères : Dr Vincent Ecochard, IPBS (Toulouse)
- Détecteurs bio-puces : Dr Laurence Salomé, IPBS (Toulouse)
Publications
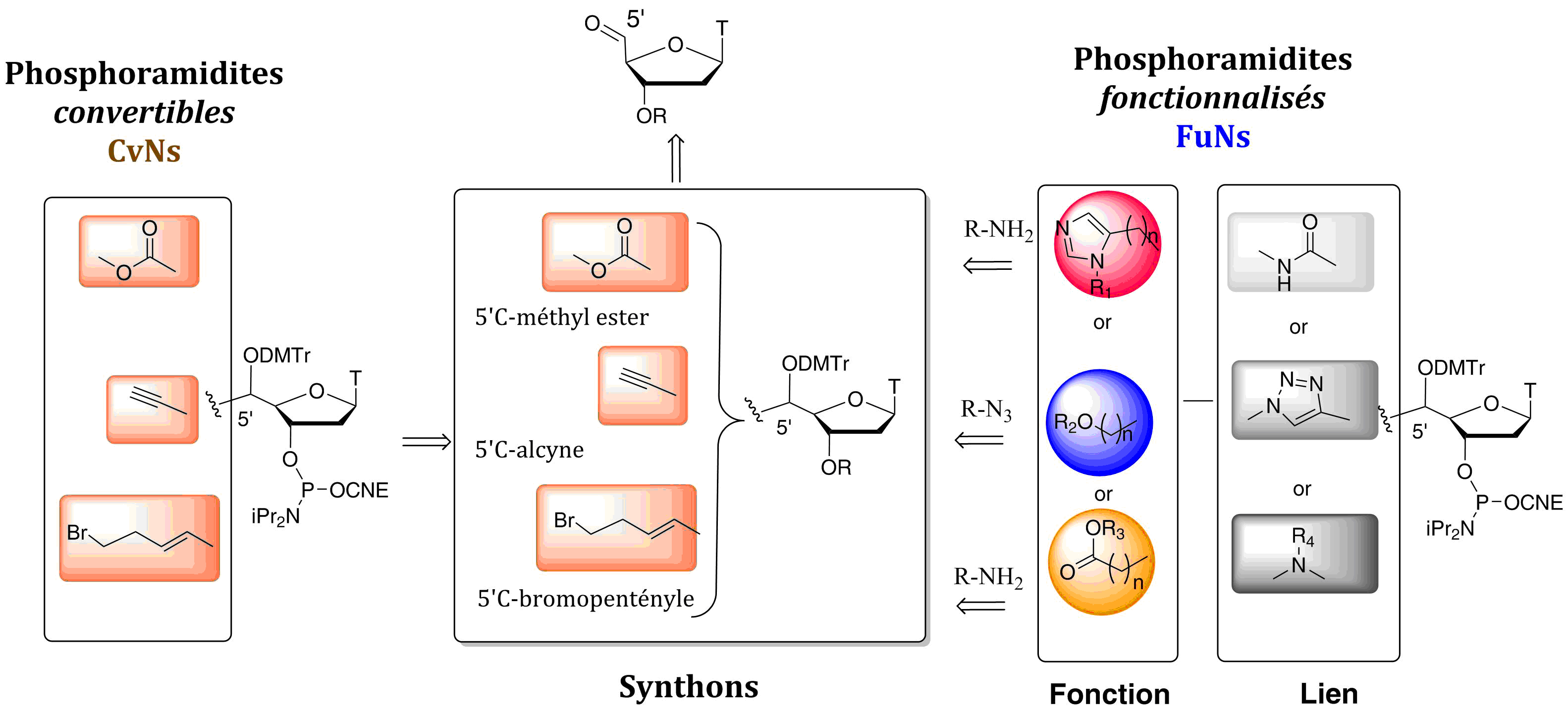
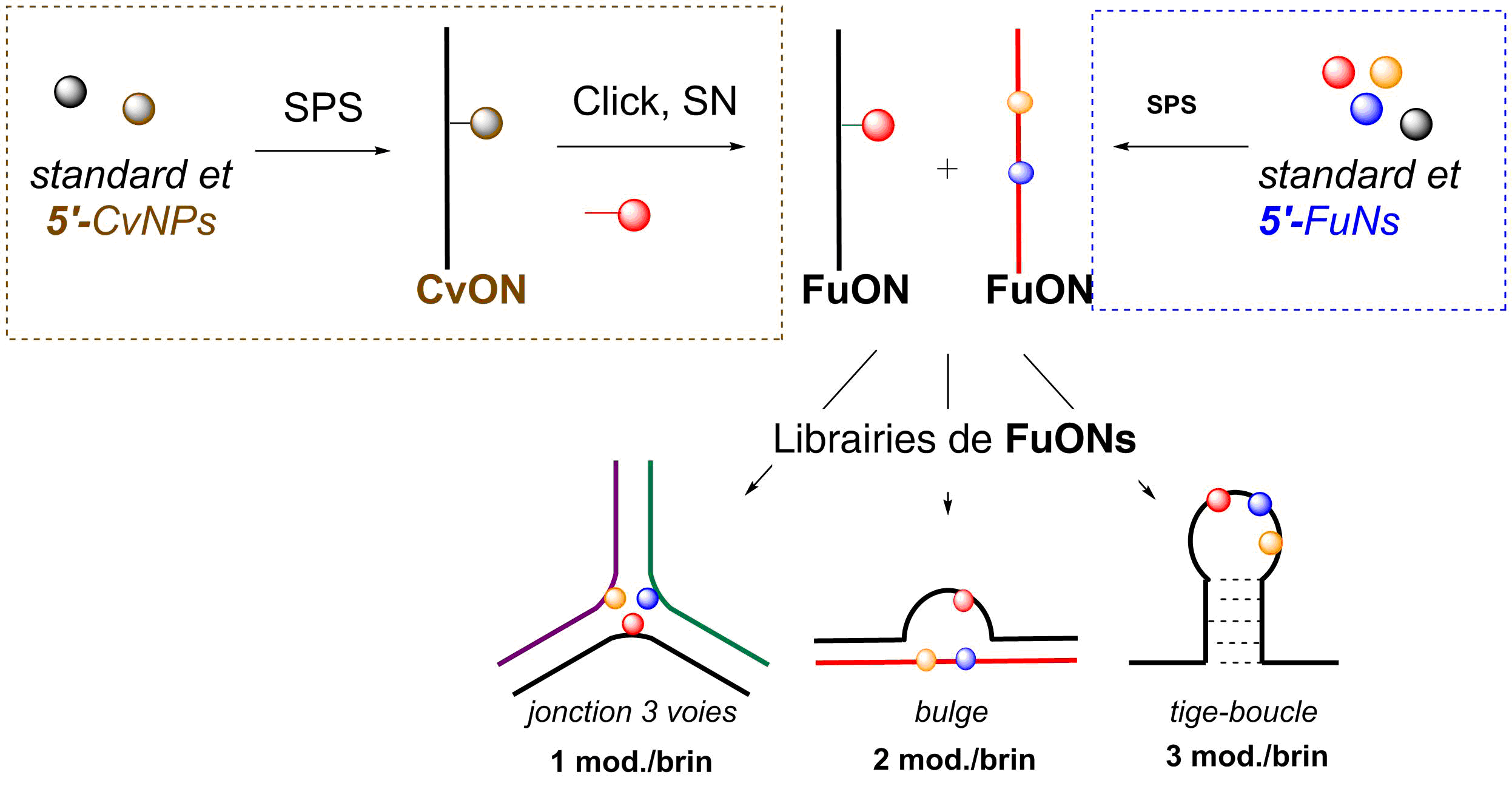
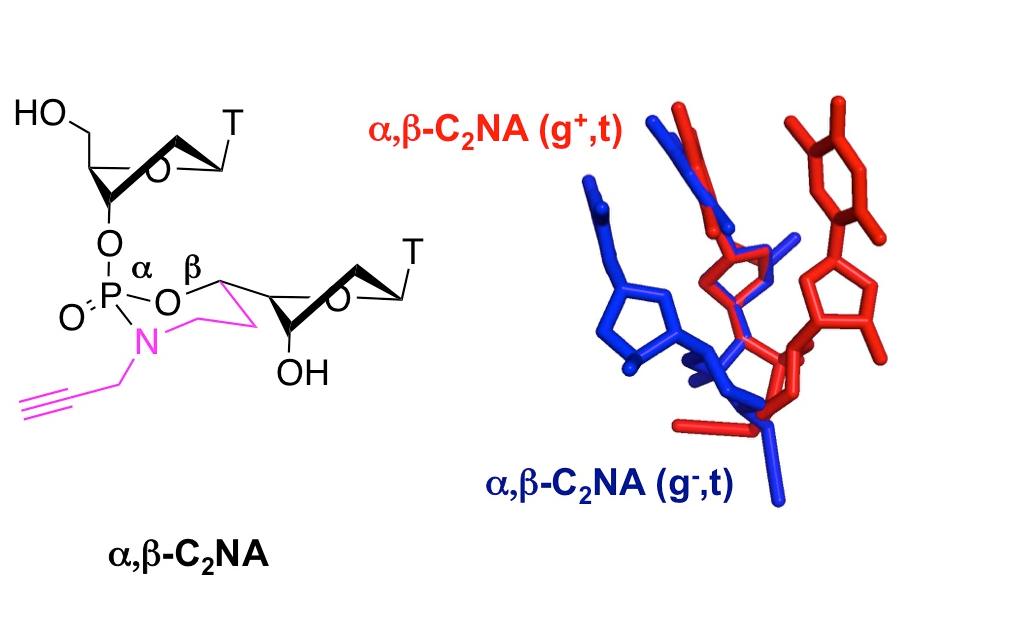
- Escudier, J.-M., Payrastre, C., Gerland, B., and Tarrat, N. Convertible and conformationally constrained nucleic acids (C2NAs), (2019), Org. Biomol. Chem. 17, 6386-6397. Voir la publication
- Gerland, B., Addamiano, C., Renard, B.-L., Payrastre, C., Gopaul, D., and Escudier, J.-M. Thio- and Seleno-Dioxaphosphorinane-Constrained Dinucleotides (D-CNA): Synthesis and Conformational Study, (2017), Eur. J. Org. Chem. 1450-1464. Voir la publication
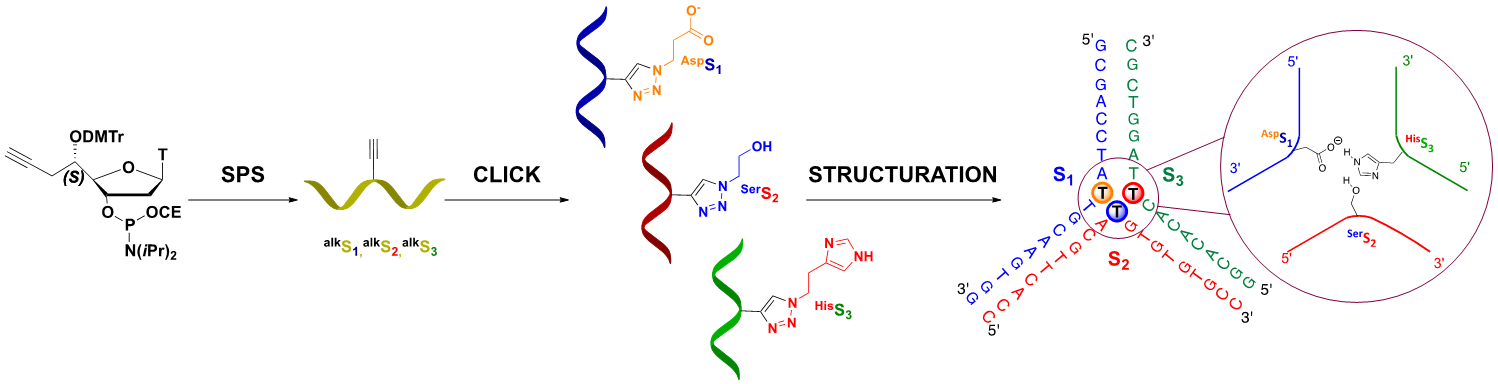
- Addamiano, C., Gerland, B., Payrastre, C., and Escudier, J. M. DNA Three Way Junction Core Decorated with Amino Acids-Like Residues-Synthesis and Characterization, (2016), Molecules 21, 1082. Voir la publication
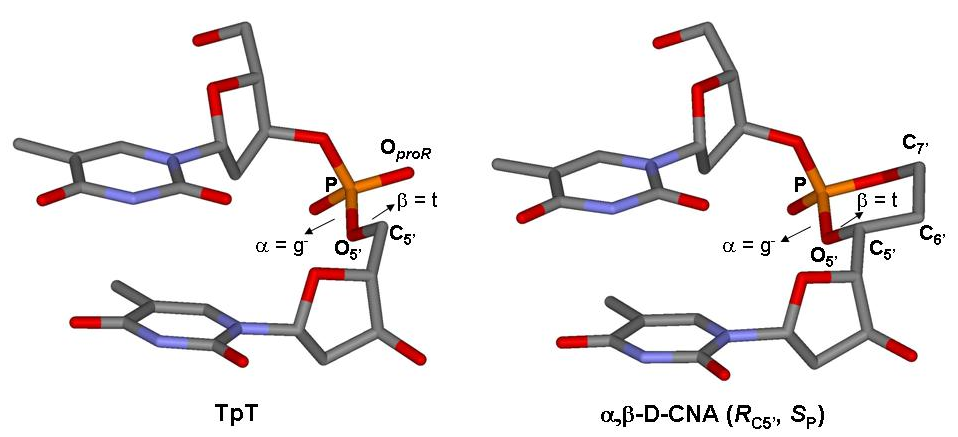
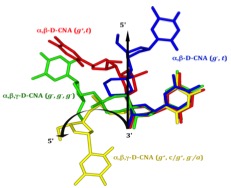
- Gerland, B., Millard, P., Dupouy, C., Renard, B.-L., and Escudier, J.-M. Stabilization of hairpins and bulged secondary structures of nucleic acids by single incorporation of α,β-D-CNA featuring a gauche(+) alpha torsional angle, (2014), RSC Advances 4, 48821-48826. Voir la publication
- Østergaard, M. E., Gerland, B., Escudier, J.-M., Swayze, E. E., and Seth, P. P. Differential Effects on Allele Selective Silencing of Mutant Huntingtin by Two Stereoisomers of α,β-Constrained Nucleic Acid, (2014), ACS Chemical Biology 9, 1975-1979. Voir la publication
- Maether, M.-P., Lapin, K., Muntean, A., Payrastre, C., and Escudier, J.-M. Oligonucleotide Labelling Using a Fluorogenic “Click” Reaction with a Hemicarboxonium Salt, (2013), Molecules 18, 12966-12976. Voir la publication
Financement
- ANR Blanc Modulation of Integrase Activity by Means of Conformationaly Constrained Nucleotides (MIAM CoCoNuts). (2011).
- ANR JCJC Protease-like oligonucleotides, functionalization and catalysis (PrOLIFiC). (2018).